Gráficas Analíticas 2D
Para comenzar utilizaremos el comando más básico para graficación. Igualmente cargaremos en memoria una biblioteca
de comandos gráficos
El comando plot
Generalidades de plot
Hay varias formas de contruir una función
| > | f :=(x) -> x*sin(x); |
![]()
y se grafica
| > | plot(f(x), x = -99..99); |
![[Plot]](visualizacionimag/visualizacion_2.gif)
nótese el juego con los botones de arriba.
Igual se puede graficar la función directamente y seleccionar una visión particular
| > | plot(x*sin(x), x = -10..10, y=0..4); |
![[Plot]](visualizacionimag/visualizacion_3.gif)
La Sintaxis
La sintaxis del comando plot es la siguiente
plot(funcion-datos, horiz, vert, opciones)
All but the first argument are optional, but arguments must be given in the order shown above.
funcion-datos: expresión, mapping, lista o conjunto de datos a graficar
horiz / vert:
variable: etiquetas y ejes
rang0: determina el rango de graficación
variable = rango: ambos
opciones: ecuaciones de la forma opción = VALOR
Para encontrar la lista de opciones se busca ?plot[options]
Warning, inserted missing semicolon at end of statement, ...rt, opciones);
Error, (in plot) invalid arguments
Ejemplos, escalas y etiquetas
| > | plot(tan(x), x=-Pi..Pi); |
![[Plot]](visualizacionimag/visualizacion_4.gif)
la escala vertical afecta la visión, para restringir la escala podemos proceder
| > | plot(tan(x), x=-Pi..Pi, `tan(x)`=-5..5); # notese el entrecomillado inverso, ` `, para el eje vertical |
![[Plot]](visualizacionimag/visualizacion_5.gif)
Las comillas inversas `, se utilizan para encerrar textos que quisiéramos que se desplieguen en la gráfica.
nótese el símbolo de # que inhibe cualquier comando que lo siga.
Adicionalmente, vemos que las líneas verticales son espúrias. Para ello le podemos indicar al comando plot que la
función es discontínua
| > | plot(tan(x), x=-Pi..Pi, `tan(x)`=-5..5,discont=true,xtickmarks=[-3.14=`-Pi`,-1.57=`-Pi/2`,1.57=`Pi/2`,3.14=`Pi`]); |
![[Plot]](visualizacionimag/visualizacion_6.gif)
Incluimos en este comando, una manera más intuitiva de ver el eje x. En el eje y, las etiquetas se puenden voltear y el título de la grafica se puede colocar, si nos ponemos un poco más exquisitos.
| > | plot(tan(x), x=-Pi..Pi, `tan(x)`=-5..5,discont=true,xtickmarks=[-3.14=`-Pi`,-1.57=`-Pi/2`,1.57=`Pi/2`,3.14=`Pi`],
labels=[`x`,`tan(x)`],title=`tan(x) vs x`,labeldirections = [horizontal, vertical]); |
![[Plot]](visualizacionimag/visualizacion_7.gif)
Adicionalmente, también podemos explorar la función
| > | g:=(x)->sin(x^2)/x^2; |
![]()
| > | plot(g(x),x=-2*Pi..2*Pi); |
![[Plot]](visualizacionimag/visualizacion_9.gif)
y notamos que los ejes no tienen la misma escala, por lo cual nuestra intuición se puede ver traicionada, entonces a veces
es bueno colocar scaling=constrained
| > | plot(g(x),x=-2*Pi..2*Pi, `sin(x^2)/x^2.`=-0.5..1.5,
xtickmarks=[-6.28=`-2Pi`,-4.71=`-3Pi/2`,-3.14=`-Pi`,-1.57=`-Pi/2`, 1.57=`Pi/2`,3.14=`Pi`,4.71=`3Pi/2`,6.28=`2Pi`],scaling=constrained); |
![[Plot]](visualizacionimag/visualizacion_10.gif)
Algunas veces las funciones son razonables y no evolucionan muy rápido
| > | plot(g(x),x=-infinity..infinity, `sin(x^2)/x^2.`=-0.5..1.5, scaling=constrained); |
![[Plot]](visualizacionimag/visualizacion_11.gif)
Asignación y manipulación de gráficas
Las gráficas se pueden asignar a variables para luego almacenarlas, imprimirlas o mostrarlas en pantalla. En ese caso para no ser testigo de la asignación de un inmenso y casi inenteligible conjunto de números, debemos terminar la asignación con dos punto :
| > | graftan:=plot(tan(x), x=-Pi..Pi,
`tan(x)`=-5..5,discont=true,xtickmarks=[-3.14=`-Pi`,-1.57=`-Pi/2`,1.57=`Pi/2`,3.14=`Pi`]): |
luego podremos mostrar la gráfrica con solo invocar la varible
| > | graftan; |
![[Plot]](visualizacionimag/visualizacion_12.gif)
Podemos almacenar la gráfica para utilizarla en este u otra de maple
| > | save graftan, "c:/Luisn/Latex/cursos/visualizacion/graftan.m": |
Nótese la forma de escribir el trayecto para almacenar el archivo y que el nombre del archivo debe tener la extensión .m para que MAPLE escriba el nombre de la variable y su asiganción en el formato interno de MAPLE. De esta forma el archivo es recuperable y se puede incluir en cualquier otra hoja de trabajo.
| > | graftan := NULL: # con esta expresion "aNULLamos" el valor de la variable para luego asignarle otro
graftan; read "c:/Luisn/Latex/cursos/visualizacion/graftan.m": graftan; |
![[Plot]](visualizacionimag/visualizacion_13.gif)
Como el archivo graftan.m contiene el nombre de la variable y su asignación, nos vimos abligados a "limpiar" el contenido de la variable antes de leerla del archivo donde fue almacenada. Esta acción no será necesaria si leemos esta gráfica desde otra hoja de trabajo MAPLE.
Disgresión en la estructura
Tipos de Datos, listas, conjuntos .....y ¿ qué más
La visualización de los datos simpre impone conocer algo de la estructura con la cual estamos trabajando. Conocer los datos Y su estructura es vital para su representación gráfica. Exploraremos aquí algunas particularidades de la representación de datos utilizando esta herramienta. MAPLE dispone de diversas maneras de representar los datos. Veamos el siguiente ejemplo con el comando solve(eqn, var) que implica resolver una ecuación algebráica, eqn, para una de las variables involucradas var
| > | polinomio:=x^4-1; # shif + enter nos deja dentro del mismo grupo de comandos
solve(polinomio, x); |
![]()
![]()
MAPLE nos devuelve una secuencia de soluciones. Ahora bien, si procedemos a especificar UNA de las soluciones, tendríamos que
| > | solve(polinomio, x)[1]; |
![]()
| > | argumento:=polinonimo,x; |
![]()
| > | whattype(argumento); |
![]()
claramente es una expresion de una secuencia !
Primeramente, diremos que utilizaremos de manera indistinta la palabra dato abstracto u objeto. En segundo lugar puntualizaremos que indicaremos estos objetos de distinto modo: mediante series de objetos separados por comas entre paréntisis (), objetos separados por comas entre llaves {} u objetos separados por comas entre corchetes []. Cada uno de estos conjuntos de objetos representa una estructura de datos distinta y tendrá una representación, utilización y un álgebra distinta. Nótese que ya hemos utilizado algunas de estas estructuras. Además de los argumentos de las funciones (objetos separados por comas dentro de paréntesis) en el caso de las etiquetas (labels=[,])
Consideraremos las siguientes estructuras de datos para graficar
Secuencias: Es el conjunto de datos u objetos separados por comas. Los argumentos de las funciones en MAPLE son, de acuerdo con esta definición, secuencias.
Conjuntos: Recuerda a la estructura y operación de los conjuntos matemáticos: es un conjunto de elementos separados por comas y encerrados entre llaves en los cuales los elementos aparecen un única vez y el orden de los elementos no importa. Pudiera decirse que un conjunto es una secuencia entre llaves.
Listas: Si un conjunto era una secuencia encerrada entre llaves, diremos ahora que una lista es un conjunto de conjuntos separados por comas encerrados entre corchetes, pero tambien es una secuencia encerrada entre llaves. A diferencia de los conjuntos el orden (y la posición) en el cual aparecen los elementos de la lista DEFINITIVAMENTE SI importa. Además los elementos pueden aparecer repetidos.
Arreglos: Este tipo de estructura de datos corresponde con la representación de vectores y matrices y en el caso de visualización utilizaremos las matrices para graficar archivos de datos experimentales
Tabla: La mencionamos por completitud, pero no abundaremos en su significado y uso, sólo diremos que es una estructura similar a la de registro (record) en lenguajes como Pascal o C.
Así, podemos ejemplificar lo anteriormente expuesto
| > | ecua1:=3*x +4*y=5;
ecua2:=8*x -y=16; solve( {ecua1, ecua2}, {x, y}); |
![]()
![]()
![]()
constituye el conjunto de soluciones, el cual, obviamente es equivalente a
| > | solve( {ecua2, ecua1}, {y, x}); |
![]()
y en contraposición al ejemplo anterior, si invertimos el orden de una lista, el resultado es otro
| > | plot(x*sin(x),x =-10..10,labels=[`f(x)`,`x`],title=`f(x) vs x`); |
![[Plot]](visualizacionimag/visualizacion_23.gif)
Finalmente, podremos representar un vector como
| > | v:=array( [1+a,3+2*a +b,8+3*a+5*b+8*c]); |
![]()
y verificarlo
| > | type(v,list); |
![]()
| > | type(v,vector); |
![]()
| > | type(v,array); |
![]()
una matriz
| > | M:=array([ [1-p,2-q],[2-p*q,3-p^q] ]); |
![M := matrix([[1-p, 2-q], [2-p*q, 3-p^q]])](visualizacionimag/visualizacion_28.gif)
Un mínimo de Operaciones con la estructura de datos
Operadores
| > |
Operaciones
| > |
Varias curvas en una gráfica
Para representar varias curvas en una gráfica utilizamos un conjunto de curvas. Esto es le dato abstracto conjunto por cuanto no repetiremos una curva y su orden no nos importará. De este modo, tendremos un conjunto como primer objeto de la secuencia que es el argumento de la función
| > | plot({sin(wt),-cos(wt),-sin(wt)}, wt = -Pi..1.5*Pi); |
![[Plot]](visualizacionimag/visualizacion_29.gif)
o acomodando un poco esta gráfica
| > | plot({sin(wt),-cos(wt),-sin(wt)}, wt = -Pi..1.5*Pi,
scaling=constrained, title = "Posición, Velocidad y Aceleración", axes = BOXED,colour = [orange, blue,green]); |
![[Plot]](visualizacionimag/visualizacion_30.gif)
Pero no podemos colocar una leyenda para diferenciar curvas porque hemos graficado un conjunto de funciones y no una lista de funciones. Si queremos incluir una leyenda que diferencie las curvas en la gráficas, tenemos que graficar una lista de funciones donde el orden de las curvas importe.
| > | plot([sin(wt),-cos(wt),-sin(wt)], wt = -Pi..1.5*Pi,
scaling=constrained, title = "Posición, Velocidad y Aceleración", axes = BOXED, colour = [orange, blue,green], legend = ["Posición", "Velocidad","Aceleración"]); |
![[Plot]](visualizacionimag/visualizacion_31.gif)
Pero también podremos representar el típo y el grosor de la línea para cuando no tengamos una impresión en colores
| > | plot([sin(wt),-cos(wt),-sin(wt),wt], wt = -Pi..1.5*Pi,
scaling=constrained, title = "Posición, Velocidad y Aceleración", axes = normal, colour = [orange, blue,green,black], legend = ["Posición", "Velocidad","Aceleración","argumento"], linestyle=[1,2,3,4], thickness=[3,2,1,0]); |
![[Plot]](visualizacionimag/visualizacion_32.gif)
y todo lo anterior se puede cambiar utilizando las barra de herramientas que provee MAPLE cuando se marca la gráfica.
Explorando Curvas
Podemos trambien delimitar una ventana de visualizacion con la opción view= [xmax..xmin,ymax..ymin] EL RANGO DE Y
| > | plot([sin(x),tan(x),x], x = 0..Pi, view=[0..Pi, -Pi..Pi],scaling=constrained,discont=true); |
![[Plot]](visualizacionimag/visualizacion_33.gif)
o también
| > | plot([sin(x),tan(x),x], x=0..Pi,view=[0..1.5, -2..2],scaling=constrained,discont=true); |
![[Plot]](visualizacionimag/visualizacion_34.gif)
| > | plot([sin(x),min(10,tan(x)), x], x = -Pi..Pi,discont=true); |
![[Plot]](visualizacionimag/visualizacion_35.gif)
| > | plot([sin(x),max(-10,tan(x)), x], x = -Pi..Pi); |
![[Plot]](visualizacionimag/visualizacion_36.gif)
| > | plot([sin(x),max(-5, min( 5, tan(x) ) ), x], x=-Pi..Pi,scaling=constrained,discont=true); |
![[Plot]](visualizacionimag/visualizacion_37.gif)
Variedades de Curvas
En los puntos anteriores hemos explorado la forma estándar de curva, esto es y(x) vs x, ahora trataremos de
explorar otros tipos
Paramétricas
Típicamente hemos visto curvas f(x) vs x y hemos graficado varias curvas en una misma gráfica f(t) vs t y g(t) vs t . Con ello lo que queremos decir es que graficamos el lugar geométrico tal que (x = t, y=f(t)) y ( x = t, y=g(t)) Ahora la idea es graficar ambas curvas etiquetadas por ese parámetro t tal que (x=f(t), y=g(t) ).Por ejemplo graficamos el lugar geométrico de todos los puntos (t^2,t) tendremos que la sintaxis será clara, plot([x(t), y(t), t=rango de t], h, v, opciones) los dos primeros elementos de la lista del argumento seran x, y, variación del parámetro
| > | plot([t^2, t, t = -1..1]); |
![[Plot]](visualizacionimag/visualizacion_38.gif)
igualemente
| > | plot([sin(t^2), t, t = -2*Pi..2*Pi]); |
![[Plot]](visualizacionimag/visualizacion_39.gif)
En Física cuando graficamos la velocidad vs la posición llamamos a ese espacio el espacio de fases. Así para un oscilador armónico simple tendremos
| > | omega0:=2*Pi/5:
xosc(t):= 2*sin(omega0*t); plot( [xosc(t), diff(xosc(t),t), t = -5..5], labels=[`Posicion`,`Velocidad`],labeldirections = [horizontal, vertical]); |
![]()
![[Plot]](visualizacionimag/visualizacion_41.gif)
Quizá el ejemplo, más emblemático de representación paramétrica de una curva lo constituye la circunsferencia centrada en el origen que viene dada por el lugar geométrico, coordenadas cartesianas, de los puntos (x,y) -> ![]()
![]() , entonces pod
, entonces pod
| > | radio1 := 5:
plot([radio1*cos(t), radio1*sin(t), t = 0..2*Pi], scaling = CONSTRAINED); |
![[Plot]](visualizacionimag/visualizacion_44.gif)
Polares
Una gráfica en coordenadas polares viene dada por la representación de![]() Si consideramos un radio constante y graficamos su trayectoria variando el ángulo
Si consideramos un radio constante y graficamos su trayectoria variando el ángulo ![]() describimos una circunsferencia centrada en el origen. Es equivalente a graficar una curva paramétrica del lugar geométrico del tipo (x=
describimos una circunsferencia centrada en el origen. Es equivalente a graficar una curva paramétrica del lugar geométrico del tipo (x= ![]() , y=
, y= ![]() ) y se varía el parámetro
) y se varía el parámetro ![]() obviamente le indicamos a MAPLE que la gráfica viene en coordenadas plares. Si graficamos la misma circunsferencia centrada en el origen del punto anterior tendremos:
obviamente le indicamos a MAPLE que la gráfica viene en coordenadas plares. Si graficamos la misma circunsferencia centrada en el origen del punto anterior tendremos:
| > | radio2 :=5;
plot([radio2 , theta, theta=0..2*Pi],coords=polar); |
![]()
![[Plot]](visualizacionimag/visualizacion_51.gif)
si por el contrario ![]() entonces
entonces
| > | plot([sin(theta) , theta, theta=0..2*Pi],coords=polar, scaling=constrained); |
![[Plot]](visualizacionimag/visualizacion_53.gif)
En la Aproximación de Born para dispersión elástica de electrones que inciden sobre átomos, se obtiene la expresión para la sección eficaz diferencial para el scatering apantallado de Rutherford.
| > | sigma1:=(k,theta)->(1/4)*( (Z^2*(1-(1/(1+4*k^2*a^2*sin(theta/2)^2)^2))^2)/(k^4*sin(theta/2)^4)); |
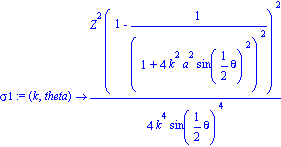
en la cual ![]() es la energía de dispersión que se considera fija; Z es el número atómico y a corresponde con el factor de apantallamiento. Si graficamos la sección eficaz diferencial respecto al ángulo, nos queda algo como
es la energía de dispersión que se considera fija; Z es el número atómico y a corresponde con el factor de apantallamiento. Si graficamos la sección eficaz diferencial respecto al ángulo, nos queda algo como
| > | plot(subs(a=1/2,Z=1,sigma1(1,theta)), theta=0..2*Pi, labels=["theta","sigma"]); |
![[Plot]](visualizacionimag/visualizacion_56.gif)
es mucho más intuitiva si realizamos la gráfica en coordenadas polares.
| > | plot([subs(a=1/2,Z=1,sigma1(1,theta)), theta, theta=0..2*Pi], coords= polar,scaling=constrained); |
![[Plot]](visualizacionimag/visualizacion_57.gif)
Es mucho más evidente el patrón de dispersión el valor de la función para cada ángulo. Algo parecido presenta R. Landau en el patrón de dispersión de Rayos x de baja energía dispersados por una esfera
| > | sigma:=3+2*cos(theta)^4 + 2*cos(theta);
plot(sigma, theta=0..2*Pi, labels=["theta","sigma"], scaling=constrained); |
![]()
![[Plot]](visualizacionimag/visualizacion_59.gif)
no es obvia la forma del patrón de dispersión cosa que es clara cuando se grafica en término de coordenadas polares
| > | plot([sigma, theta, theta=0..2*Pi],coords=polar,scaling=constrained); |
![[Plot]](visualizacionimag/visualizacion_60.gif)
El Paquete plots
El paquete plots aumenta las posibilidades de graficación al incluir un conjunto de "comandos" adicionales. Las funciones del plots se pueden invocar directamente haciendo referencia al paquete plots. Esto es: plots[setoptions](scaling = CONSTRAINED). Lo más común es que se utilicen directamente como setoptions(scaling = CONSTRAINED), luego de incorporar el paquete completo de funciones como lo es usual en MAPLE a través del comando
| > | restart;with(plots): |
Warning, the name changecoords has been redefined
Los nombres que arriba aparecen hacen intuir la funcionalidad del comando. Mostraremos el uso de algunas de las opciones y dejaremos al lector explorar algunas de las otras mediante la utilización de la ayuda en línea.
Cambio de opciones por omisión
Lo primero que exploramos en el uso de los nuevos comandos que provee el paquete plots es la posibilidad de crear un conjunto, personalizado de opciones por omisión. Así las opciones por omisión pueden ser alteradas mediante
| > | setoptions(scaling = CONSTRAINED); |
Opción que será supuesta por omisión a partir de este momento y la cual pude ser revertida mediante:
setoptions(scaling = UNCONSTRAINED);
Gráficas Logarítmicas
Seguidamente exploraremos otra de las opciones de plots que permite realizar gráficas logarítmicas, mediante los comandos logplot (eje vertical logarítmico), semilogplot (eje horizontal logarítmico) loglogplot (ambos ejes logarítmicos)
Así tendremos que
| > | semilogplot([ln(3*x)/10,log[10](x)], x=1..2*Pi,
labels=["log10(x)","y"],labeldirections = [horizontal, vertical],legend = ["ln(3*x)/10","log[10](x)"], title="Grafica Semi log = eje x log"); |
![[Plot]](visualizacionimag/visualizacion_61.gif)
| > | logplot([ln(3*x)/10,log[10](x)], x=1..2*Pi,
labels=["x","log10(y)"],labeldirections = [horizontal, vertical],legend = ["cos(x) + sen(x)","log[10](x)"], title="Grafica Semi log = eje y log",numpoints=500); |
![[Plot]](visualizacionimag/visualizacion_62.gif)
Nótese como hemos aumentado el número de puntos de graficación a través de la opción numpoints=500
| > | loglogplot([ln(3*x)/10,log[10](x)], x=1..2*Pi,
labels=["log10(x)","log10(y)"],labeldirections = [horizontal, vertical],legend = ["cos(x) + sen(x)","log[10](x)"], title="Grafica Semi log = ejes x y log",numpoints=500 ); |
![[Plot]](visualizacionimag/visualizacion_63.gif)
| > |
Otra vez, varias Gráficas en una: la función display
Otra forma de representar varias curvas en una sola gráfica es a través de la función display del paquete plots. Esta función permite mostrar varias gráficas mediante la asignación de gráficas a variables, para luego "mostrar" el conjunto de variables. Así el ejemplo más trivial lo constituye las g[raficas del seno y el conseno. Esto es
| > | S := plot(sin): C := plot(cos):
display({S,C}, title = "Seno y Coseno"); |
![[Plot]](visualizacionimag/visualizacion_64.gif)
Un ejemplo más interesante lo constituye el movimiento bajo fuerzas centrales cuando consideramos un potencial del tipo ![]() el cual es típico en el movimiento planetario o en movimentos bajo fuerzas coulombinas. Es claro que
el cual es típico en el movimiento planetario o en movimentos bajo fuerzas coulombinas. Es claro que ![]() para el caso de gravitación universal newtoniana o
para el caso de gravitación universal newtoniana o ![]() para el caso de las fuerzas coulombianas. Para este caso la trayectoria de los cuerpos sometidos a la acción de la fuerza que deriva del potencial será
para el caso de las fuerzas coulombianas. Para este caso la trayectoria de los cuerpos sometidos a la acción de la fuerza que deriva del potencial será
| > | r1:=(theta)->lambda*(1+epsilon)/(1+epsilon*cos(theta-theta0)); |
![]()
donde 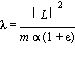 y
y ![]() , la excentricidad, viene dado por
, la excentricidad, viene dado por 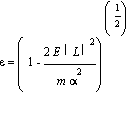 Con L la cantidad de movimiento angular; E la energía total. Nóteses que
Con L la cantidad de movimiento angular; E la energía total. Nóteses que ![]() será positivo, para potenciales atractivos (
será positivo, para potenciales atractivos (![]() > 0 ) y será negativo para potenciales repulsivos (
> 0 ) y será negativo para potenciales repulsivos ( ![]() < 0). Igualmente recordemos que la esta evolución corresponde a una representacion de secciones cónicas y las cuales se ilustran
< 0). Igualmente recordemos que la esta evolución corresponde a una representacion de secciones cónicas y las cuales se ilustran
| > | theta0:=0;
rElipse := subs (lambda=1,epsilon=1/2,r1(theta)): rParabola := subs (lambda=1,epsilon=1,r1(theta)): rHiperbolaP := subs (lambda=1,epsilon=2,r1(theta)): rHiperbolaN := subs (lambda=-1,epsilon=2,r1(theta)): grfrElipse:=plot([rElipse,theta,theta=0..2*Pi],coords=polar, color=red): grfrParabola:=plot([rParabola,theta,theta=0..2*Pi],coords=polar, color =green ): grfrHiperbolaP:=plot([rHiperbolaP,theta,theta=0..2*Pi],coords=polar, color =blue ): grfrHiperbolaN:=plot([rHiperbolaN,theta,theta=0..2*Pi],coords=polar,color =yellow): display([grfrElipse,grfrParabola,grfrHiperbolaP,grfrHiperbolaN], view=[-10..1,-10..10], title="Secciones Cónicas"); |
![]()
![[Plot]](visualizacionimag/visualizacion_76.gif)
Explorando el comportamiento de datos con animaciones.
Existen dos formas de representar animaciones con MAPLE: una limitada utilizando el comando animate y otra forma utilzando el
comando display analicemos ambos casos
Animaciones con animate
La sintaxis del comando animate es animate(pcomandoplot, argplot, t=a..b,...) Este comado genera una gráfica y al seleccionarla aparecen botones parecidos a los controles de una videograbadora.
Consideremos la solución sub-amortiguada del oscilador libre, amortiguado
| > | restart;with(plots):
xamort(t) := 1/2*(mu*x0+(mu^2-omega0^2)^(1/2)*x0+v0)/(mu^2-omega0^2)^(1/2)*exp((-mu+(mu^2-omega0^2)^(1/2))*t)-1/2*(mu*x0-(mu^2-omega0^2)^(1/2)*x0+v0)/(mu^2-omega0^2)^(1/2)*exp((-mu-(mu^2-omega0^2)^(1/2))*t); |
Warning, the name changecoords has been redefined
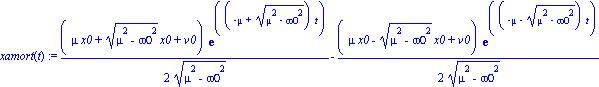
Para analizar su comportamiento bajo variación de parámetros, podemos considerar un par de formas de animar esa gráfica.
Primeramente, consideremos variaciones de la velocidad inicial para un coeficiente de amortiguamiento, una masa y una constante
elástica dada. Esto es
| > | paramV:=[x0=0,mu=0.5,omega0=2]:Xv(t):=subs(paramV,xamort(t));
animate(plot,[Xv(t),t=0..10],v0=1..10); |
![]()
![[Plot]](visualizacionimag/visualizacion_79.gif)
También podemos explorar el comportamiento bajo variacioes del coeficiente de amortiguamiento, estos es
| > | parammu:=[x0=0,v0=5,omega0=2]:Xmu(t):=subs(parammu,xamort(t));
animate(plot,[Xmu(t),t=0..10],mu=0.1..1.99, frames=100); |
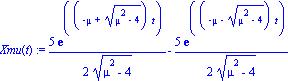
![[Plot]](visualizacionimag/visualizacion_81.gif)
También se pueden superponer una animación con un fondo
| > | ondaseno := plot( sin(x)*exp(-x/5),x=0..20 ):
animate( pointplot, [ [[t,sin(t)*exp(-t/5)]],symbol=circle,symbolsize=10], t=0..20, frames=60, background=ondaseno ); |
![[Plot]](visualizacionimag/visualizacion_82.gif)
y comparar la evolución de una curva respecto a otra
| > | curva := implicitplot( x^3+y^2, x=-3..1, y=-4..4, color=blue ):
animate( implicitplot, [x^3-A*y+y^2,x=-3..1,y=-4..4], A=-2..2, background=curve, frames=50 ); |
Error, (in animate) background value must be a real number or a plot structure
| > |
Animaciones utilizando display
Con la opción insequence = true, display combina una lista gráficas que cambian con el tiempo así, la gráfica (paramétrica ) de una línea recta
| > | setoptions(scaling = CONSTRAINED);
plot([[0,0], [cos(Pi/4),sin(Pi/4)]], 0..1, 0..1); |
![[Plot]](visualizacionimag/visualizacion_83.gif)
Animémosla con una secuencia de 16 cuadros con ![]() evolucionando de 0 con incrementos de
evolucionando de 0 con incrementos de ![]() . Primeramente, construimos la secuencia de gráficas
. Primeramente, construimos la secuencia de gráficas
| > | plot_seq := seq(plot([[0,0], [cos(Pi/8*i),sin(Pi/8*i)]]), i = 0..15):
display([plot_seq], insequence = true); |
![[Plot]](visualizacionimag/visualizacion_86.gif)
Igual que el caso anterior, podemos animar una grárfica sobre, con otra gráfica fija.
| > | display({%, # la animacion anterior
plot([cos(t), sin(t), t = 0..2*Pi])}, # el circulo fijo title = "The hand of time"); |
![[Plot]](visualizacionimag/visualizacion_87.gif)
Campos y Contornos
Para ejemplificar una de las mejoras con el uso de las extensiones de plots supongamos dos lineas con cargas opuestas e infinitas, situadas perpendiculares al plano x,y que lo atraviezan en las posiciones (-1,0,0) y (1,0,0) el potencial electrostático vendrá dado por
| > | phi := ln(sqrt((x+1)^2)+y^2) -ln(sqrt((x-1)^2)+y^2); |
![]()
una gráfica que muestra la intensidad de estas líneas de potencial surge de
| > | gradplot(-phi,x=-2..2, y=-2..2,arrows=thick, grid=[11,11], axes=box); |
![[Plot]](visualizacionimag/visualizacion_89.gif)
Del mismo modo tendremos como gráficos de contorno
| > | contourplot(phi, x=-2..2, y=-2..2,axes=box); |
![[Plot]](visualizacionimag/visualizacion_90.gif)
con más puntos y mas contornos es mucho más clara
| > | contourplot(phi, x=-2..2, y=-2..2, numpoints=1000, contours=20); |
![[Plot]](visualizacionimag/visualizacion_91.gif)
Pero aún se puede trabajar un poco más
| > | contourplot(phi, x=-2..2, y=-2..2, numpoints=1000, contours=20,
filled=true, coloring=[white,black] ); |
![[Plot]](visualizacionimag/visualizacion_92.gif)
y si las hacemos en 3D, tendremos
| > | implicitplot3d(z=phi, x=-2..2, y=-2..2, z= -3..3); |
![[Plot]](visualizacionimag/visualizacion_93.gif)
| > | implicitplot3d(z=phi, x=-2..2, y=-2..2, z= -3..3, numpoints=5000); # con muchos más puntos |
![[Plot]](visualizacionimag/visualizacion_94.gif)
Pero podemos ir más allá y exportar estas gráficas a un formato vrml (virtual reality markup language) el cual es posible explorarlo con cualquier navegador
| > | grfcargas:=implicitplot3d(z=phi, x=-2..2, y=-2..2, z= -3..3,numpoints=500): # asigno la grafica a una variable
vrml( grfcargas, `c:/Luisn/Latex/cursos/visualizacion/cargas.wrl`): |
Curvas en el espacio
El paquete plots permite dibujar curvas en el espacio. El comando no puede ser más intuitivo plots[spacecurve]. Por su parte la sintaxis del comando es escencialmente la misma que las curvas paramétricas definidas en 2D. En general representaremos una curva en el espacio parametrizada por una variable esto es
![]()
![]()
y también podemos representarla mediante puntos que serán unidos
((![]() ), (
), (![]() ), ... ).
), ... ).
por segmentos de rectas. Así, la curva paramétrica de una hélice circular en el espacio queda representada por
| > | spacecurve([cos(t), sin(t), t, t=0..2*Pi], colour = BLACK, axes = NORMAL); |
![[Plot]](visualizacionimag/visualizacion_99.gif)
y nudos en el espacio
| > | knot:= [ -10*cos(t) - 2*cos(5*t) + 15*sin(2*t),
-15*cos(2*t) + 10*sin(t) - 2*sin(5*t), 10*cos(3*t), t= 0..2*Pi]: spacecurve(knot,axes=framed); |
![[Plot]](visualizacionimag/visualizacion_100.gif)
![[Plot]](visualizacionimag/visualizacion_101.gif)
![[Plot]](visualizacionimag/visualizacion_103.gif)
![[Plot]](visualizacionimag/visualizacion_105.gif)
![[Plot]](visualizacionimag/visualizacion_115.gif)
![[Plot]](visualizacionimag/visualizacion_122.gif)
![[Plot]](visualizacionimag/visualizacion_123.gif)
![[Plot]](visualizacionimag/visualizacion_124.gif)
![[Plot]](visualizacionimag/visualizacion_125.gif)
![[Plot]](visualizacionimag/visualizacion_126.gif)
![[Plot]](visualizacionimag/visualizacion_131.gif)
![[Plot]](visualizacionimag/visualizacion_136.gif)